基于超图的网络增强方法
作者:程嘉梁 软件学院
指导老师:高跃 软件学院
关键词:超图;网络增强;
摘要
我们直接获取的生物网络中往往充斥着大量的噪声,这些噪声会影响网络结构的准确性,对下游应用造成不利影响。针对这一问题,研究者们提出了许多解决方法,如网络增强、网络去卷积等。这些方法有一些较为明显的缺点:(1)缺乏普适性;(2)基于启发式,缺少数学解释;(3)基于简单图的低阶局部结构,信息利用率低。因此,我们提出了一种新型的生物网络优化方法:基于超图的网络增强方法(HOCNE)。该方法根据原始网络的邻接矩阵构建多超图,并从多超图中提取高阶信息反向对网络进行优化。我们在(1)GM12878细胞系中所有常染色体的1kb和5kb分辨率的Hi-C基因组网络与(2)细粒度的生物图像(蝴蝶、猴子)相似性网络中进行了实验,实验结果证明了我们的方法的有效性。
方法概述
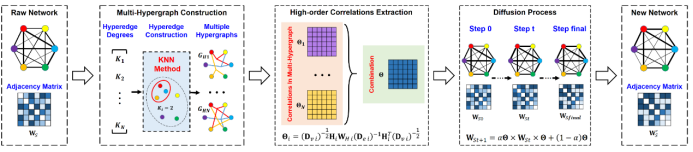
我们的方法(HOCNE)大体上可以分为5个步骤(如图1所示)。第一步,我们输入一个未经处理的有噪生物网络及其邻接矩阵。第二步,通过改变相邻节点的数量,我们使用KNN算法与输入网络的邻接矩阵构建多张超图。第三步,我们分别从每张超图中提取对应的高阶关系矩阵,并通过哈达玛积对之进行组合,并定义组合后的矩阵为多超图的高阶关系矩阵。第四步,我们将多超图高阶关系矩阵输入预定义的随机扩散过程中,通过迭代计算对网络结构进行优化。第五步,我们输出迭代最终收敛到的结果,记为我们优化后的生物网络的邻接矩阵。
对Hi-C基因组网络的优化
Hi-C是一种基于3C的技术,用于测量细胞群中成对出现的染色质相互作用的频率。Hi-C读取的数据可以构建加权无向网络,其中基因组区域是结点,两个区域之间的标准化读取映射是加权边。图2中的热力图直观地展示了我们的方法(HOCNE)卓越的性能,图3的结果说明我们的方法对应的量化指标有最大的平均值与最小的标准差,表明我们的方法更优秀也更稳定。
对细粒度生物图像相似性网络的优化
我们对蝴蝶与猴子这两类细粒度的生物图像相似性网络进行了优化,可视化结果与检索图线(图4-图7)说明我们的方法(HOCNE)可以更好地提高这一类网络的聚类特性。具体而言,HOCNE优化后网络的对应错误率较SOTA的方法NE分别降低了19%(蝴蝶网络)与63%(猴子网络)!
图片:
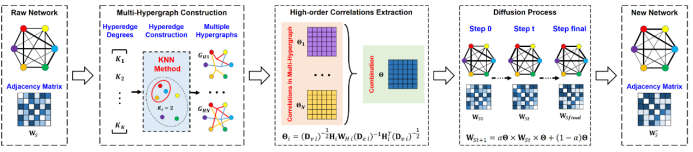
1.方法流程示意图
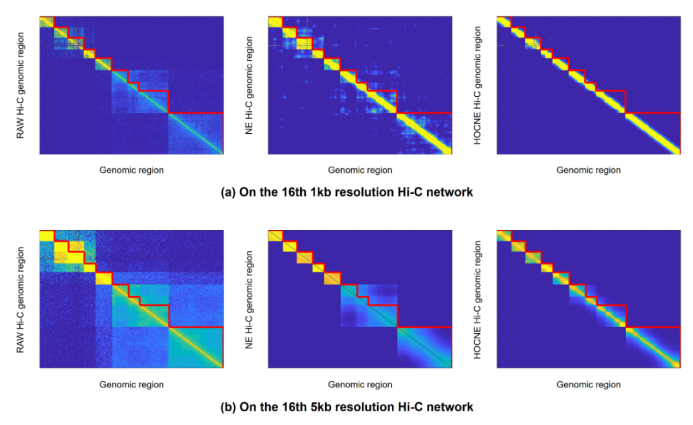
2. 16号常染色体的Hi-C关联矩阵热力图
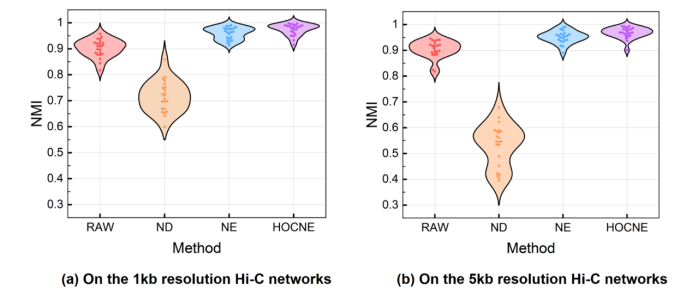
3. 所有方法量化指标对应的小提琴图

4. 蝴蝶网络的可视化
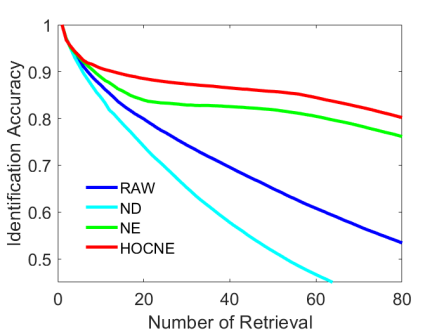
5. 蝴蝶网络的检索准确度曲线
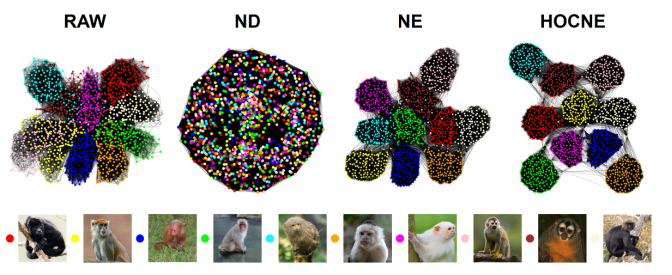
6. 猴子网络的可视化
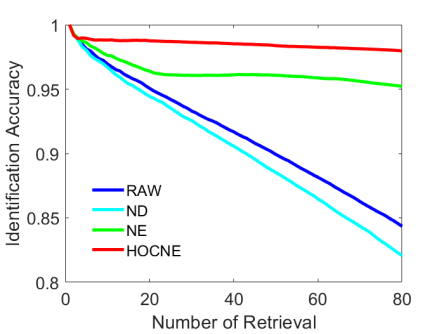
7. 猴子网络的检索准确度曲线
外部链接:邮箱:chengjl16@163.com






